Das R-Paket ForestElementsR als forstlicher Taschenrechner - Eine Schritt-für-Schritt-Einführung
von Peter Biber, Astor Toraño Caicoya und Hans-Joachim Klemmt
Die frei verfügbare Software ForestElementsR bringt Methoden der Waldmesslehre in zeitgemäßer Form auf Ihren Rechner. Das Paket bildet die technische Basis für ein langfristiges Konzept: traditionelle und alternative Ansätze zur Auswertung von Inventuren und zur Forstplanung werden sich damit flexibel umsetzen lassen. In unserem Artikel „Das R-Paket ForestElementsR – ein forstlicher Taschenrechner und mehr“ beschreiben wir den Hintergrund. Auf dieser Seite zeigen wir ein Beispiel: ForestElementsR als „forstlicher Taschenrechner“.
Hintergrund
Schritt für Schritt zum Forst-Taschenrechner
Download und Installation
Die Schritte 1-3 der folgenden Anleitung können übersprungen werden, wenn R und RStudio (empfohlen) bereits auf Ihrem Rechner installiert sind.
- Laden Sie R kostenlos von https://www.r-project.org/ herunter.
- Installieren Sie R entsprechend Ihrem Betriebssystem (Windows, macOS, Linux).
- Empfohlen: Installieren Sie zusätzlich die Benutzeroberfläche RStudio von https://posit.co/downloads/. Die kostenlose Version „RStudio Desktop“ genügt auch professionellen Ansprüchen.
- Starten Sie RStudio (bzw. R, wenn Sie RStudio nicht installiert haben) und geben Sie in die Kommandozeile ein:
R lädt das Paket von CRAN und installiert es mit allen anderen dafür benötigten Paketen, falls diese noch nicht vorhanden sind.
Einfache Operationen mit R
Wenn R gestartet ist, ist eine Eingabe an der Kommandozeile im „Taschenrechner-Stil“ der einfachste Weg, der Software Befehle zu übermitteln. Eine einfache Rechenoperation wäre wie folgt einzugeben
und würde zu folgender Antwort führen:
Möglich sind auch komplexere Eingaben; z.B. stellt der Befehl
die mathematische Funktion y=sin(x²) im Bereich −3<x<3 in roter Farbe graphisch dar:
Einzelbäume und Mitteldurchmesser
#> [1] 12.0 23.0 10.4 19.0 21.0 27.0 35.0 23.0 21.0 19.0 32.0
#> [1] 22.03636
#> [1] 23.14413
Stammdurchmesser und Winkelzählprobe
#> [1] 0.011309734 0.041547563 0.008494867 0.028352874 0.034636059 0.057255526
#> [7] 0.096211275 0.041547563 0.034636059 0.028352874 0.080424772
n_rep
#> [1] 353.67765 96.27520 470.87261 141.07917 115.48658 69.86225 41.57517
#> [8] 96.27520 115.48658 141.07917 49.73592
#> [1] 1691.406
#> [1] 18.19942
#> [1] 32.89276
d_dom_weise(bhd, n_rep)
#> [1] 26.99298
#> [1] 44
#> [1] 44
Baumarten und Stammvolumina
dg
#> [1] 18.19942
hg
#> [1] 20.01936
b_art
#> <fe_species_bavrn_state[1]>
#> [1] 60
#> [1] "Buche"
format(b_art, "eng")
#> [1] "European beech"
format(b_art, "sci")
#> [1] "Fagus sylvatica"
#> <fe_species_bavrn_state[1]>
#> [1] Buche
h
#> [1] 16.81135 21.49646 15.54876 20.30858 20.95016 22.37661 23.59196 21.49646
#> [9] 20.95016 20.30858 23.20002
#> [1] 0.08366269 0.44096199 0.05612344 0.27669387 0.35390298 0.64456520
#> [7] 1.16869636 0.44096199 0.35390298 0.27669387 0.95400515
#> [1] 441.8054
#> [1] 0.846
#> [1] 373.7674
Bonitierung und mehr
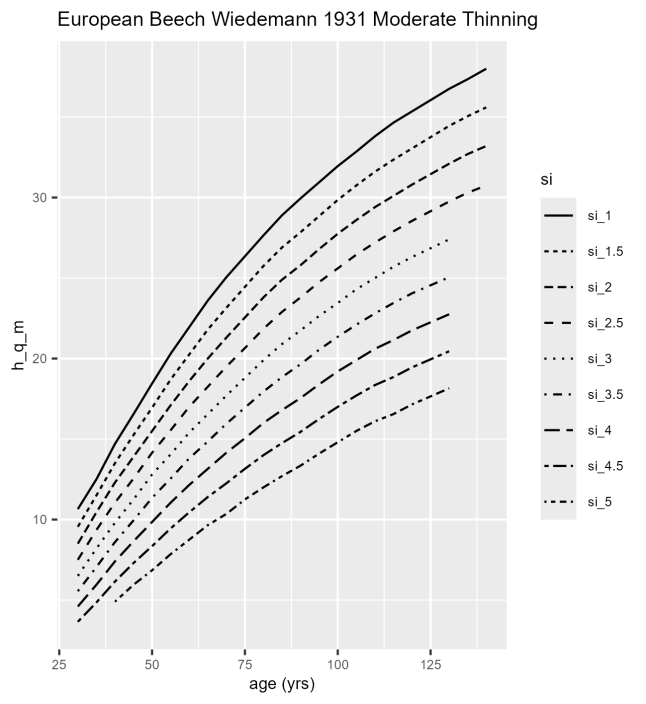
Abb. 1. Mit ForestElementsR erzeugte Darstellung des Bonitätsfächers der Ertragstafel für Rotbuche, mäßige Durchforstung, von Wiedemann (1931) (BaySTMLF 2018). Die horizontale Achse zeigt das Bestandesalter, die vertikale Achse die Höhe des Grundflächenmittelstammes. Mit „si“ ist der international übliche Begriff für die Bonität, „Site Index“ abgekürzt. Die mit „si_1.5“ bezeichnete Kurve stellt also die Höhenentwicklung bei einer Bonität von I.5 dar.
Die konkrete Bonität (international „Site Index“) des Bestandes erhalten wir mit dem Befehl site_index() unter Angabe von Alter (57 Jahre), Mittelhöhe (20.0 m) und der gewünschten Ertragstafel:
bon
#> [1] 1.292683
Der Bestand hat also eine Bonität von rund I.3; diese Information haben wir in der Variable „bon“ abgelegt. Zusammen mit dem Alter kann sie verwendet werden, um die zugehörigen Werte beliebiger Variablen aus der Ertragstafel zu ziehen. Dazu dient die Funktion ytable_lookup(), mit der wir hier den laufenden Zuwachs des Bestandes (im Paket mit der international üblichen Abkürzung „pai“ bezeichnet; „pai“ = „periodic annual increment“) schätzen wollen:
#> [1] 11.56293
Laut Ertragstafel ergibt sich so für den Bestand ein Zuwachs von rund 11,6 Festmetern pro Jahr und Hektar, s. allerdings die Anmerkungen zur Zuwachsschätzung mit Ertragstafeln von Pretzsch in BaySTMELF (2018), wo für die Buche eine Korrektur von +20% vorgeschlagen wird. Aus Gründen des Umfangs kann hier nicht weiter auf die Handhabung von Ertragstafeln mit ForestElementsR eingegangen werden. Interessierte Leser seien auf die ausführliche Darstellung in der entsprechenden Vignette des Paketes hingewiesen:
Bestandesstruktur und -diversität
Das Paket ForestElementsR ermöglicht auch erste Auswertungen zur Baumart- und Strukturdiversität von Beständen namentlich des in der Ökologie weit verbreiteten Diversitätsindex von Shannon (1948) und des Artprofilindex von Pretzsch (2019). Letzterer beruht auf dem gleichen Prinzip wie der Shannon-Index, bezieht aber auch noch die Komponente der vertikalen Bestandesstruktur ein. Zum Einsatz solcher Indizes bei der Abschätzung von Ökosystemleistungen s. Biber et al. (2020, 2021), Blattert et al. (2017) und Brockerhoff et al. (2017). Im Folgenden berechnen wir beide Indizes für unseren Beispielbestand. Hierfür ist es notwendig, für jeden einzelnen der elf Bäume die Baumart explizit anzugeben, auch wenn es sich um einen Buchenreinbestand handelt. Wir müssen also den Baumartencode 60 (s.o.) elfmal wiederholen und den erhaltenen Vektor als Baumartencodes nach dem System der Bayerischen Staatsforsten definieren:
art_vek
#> <fe_species_bavrn_state[11]>
#> [1] Buche Buche Buche Buche Buche Buche Buche Buche Buche Buche Buche
Den Shannon-Index erhalten wir mit der Funktion „shannon_index“, die neben dem Vektor mit den Artcodes auch die Repräsentationszahlen der einzelnen Bäume, „n_rep“, als Angabe benötigt:
#> [1] 0
Da wir es mit einem Reinbestand zu tun haben, ist die Baumartendiversität folgerichtig 0. Der Artprofilindex (Funktion „species_profile“) benötigt als Eingangsinformation zusätzlich noch die Höhen der Bäume (die wir zuvor in der Variable „h“ abgelegt haben):
#> [1] 0.6928344
Der erhaltene Wert ist größer als Null, weil der Bestand zwar ein Reinbestand, aber nicht völlig einschichtig ist. Zum Vergleich nehmen wir nun an, dass nicht alle unserer Bäume Buchen, sondern drei davon Eichen (Code 70) und einer davon eine Kiefer (Code 20) ist. Entsprechend definieren wir einen zweiten Vektor mit Baumartenbezeichnungen:
art_vek_2 <- as_fe_species_bavrn_state(art_vek_2)
art_vek_2
#> <fe_species_bavrn_state[11]>
#> [1] Buche Eiche (Stiel-/Trauben-) Buche
#> [4] Buche Buche Eiche (Stiel-/Trauben-)
#> [7] Kiefer Eiche (Stiel-/Trauben-) Buche
#> [10] Buche Buche
Dies führt erwartungsgemäß zu einer deutlichen Anhebung beider Indexwerte.
#> [1] 0.5426953
species_profile(art_vek_2, h, n_rep = n_rep)
#> [1] 1.096588
Im Rahmen dieser Einführung kann nicht näher auf die Funktionsweise dieser Indizes eingegangen werden; eine gut verständliche Darstellung findet sich jedoch in Pretzsch (2019). Eine wichtige Anwendung besteht darin, dass bei der Auswertung von Folgeaufnahmen durch den Vergleich dieser Indexwerte festgestellt werden kann, in welche Richtung sich die Art- und die Strukturdiversität entwickeln.
An dieser Stelle möchten wir die kurze Einführung in die Verwendung von ForestElementsR als „forstlicher Taschenrechner“ beenden. Naturgemäß konnte hier kein vollständiger Überblick über das Paket gegeben werden. Insbesondere wurde nicht auf die verschiedenen Klassen eingegangen, mit denen sich Stichprobeneinheiten oder Bestände verschiedener Definition bequem abbilden und auswerten lassen. Auch hierfür sei auf die Hauptvignette des Paketes hingewiesen.
ForestElementsR als R-Paket
- Freie Verfügbarkeit: R und die gängigste Benutzeroberfläche, RStudio, sind kostenlos.
- Standard in der Datenarbeit: R ist weltweit etabliert, von der statistischen Auswertung bis hin zur Visualisierung.
- Fester Teil der Ausbildung: Studierende in forstlichen und verwandten Studiengängen lernen fast überall Grundkenntnisse in R.
- Hervorragende Bereitstellung von R-Paketen: Nur Pakete, die strenge Standards im Hinblick auf technische Umsetzung und Dokumentation erfüllen, schaffen es in die CRAN-Bibliothek. ForestElementsR gehört dazu – frei, open source und leicht zu installieren.
- Plattformunabhängig: Alle CRAN-Pakete werden täglich auf den gängigen Betriebssystemen getestet. Eine Bindung an ein bestimmtes Betriebssystem und dessen jeweiligen Anbieter besteht deshalb für Nutzer von ForestElementsR nicht.
Literatur zur Einführung in R
Danksagung
Literaturverzeichnis
- BaySTMELF (Hrsg.), 2018, Hilfstafeln für die Forsteinrichtung. Bayerisches Staatsministerium für Ernährung, Landwirtschaft und Forsten.
- P. Biber und A. Toraño Caicoya, 2025, ForestElementsR: Data Structures and Functions for Working with Forest Data. [Online]. Verfügbar unter: https://CRAN.R-project.org/package=ForestElementsR
- P. Biber und A. Toraño Caicoya, 2025, „The Package ForestElementsR“. [Online]. Verfügbar unter: https://cran.r-project.org/web/packages/ForestElementsR/vignettes/forestelementsr_package.html
- P. Biber, 2025, „Tree Species Codings in ForestElementsR“. [Online]. Verfügbar unter: https://cran.r-project.org/web/packages/ForestElementsR/vignettes/tree_species_codings.html
- P. Biber, 2025, „Yield Tables in ForestElementsR“. 2025. [Online]. Verfügbar unter: https://cran.r-project.org/web/packages/ForestElementsR/vignettes/yield_tables.html
- P. Biber, F. Schwaiger, W. Poschenrieder, und H. Pretzsch, „A fuzzy logic-based approach for evaluating forest ecosystem service provision and biodiversity applied to a case study landscape in Southern Germany“, Eur. J. For. Res., Bd. 140, Nr. 6, S. 1559–1586, Dez. 2021, doi: 10.1007/s10342-021-01418-4.
- P. Biber et. al., 2020, „Forest Biodiversity, Carbon Sequestration, and Wood Production: Modeling Synergies and Trade-Offs for Ten Forest Landscapes Across Europe“, Front. Ecol. Evol., Bd. 8, Okt. 2020, doi: 10.3389/fevo.2020.547696.
- P. Biber et. al., 2015, „How Sensitive Are Ecosystem Services in European Forest Landscapes to Silvicultural Treatment?“, Forests, Bd. 6, Nr. 5, S. 1666–1695, Mai 2015, doi: 10.3390/f6051666.
- P. Biber, 2013, „Kontinuität durch Flexibilität – Standardisierte Datenauswertung im Rahmen eines waldwachstumskundlichen Informationssystems“, Allg. Forst- Jagdztg., Bd. 184, Nr. 7/8, S. 167–177, 2013.
- C. Blattert, R. Lemm, O. Thees, M. J. Lexer, und M. Hanewinkel, 2017, „Management of ecosystem services in mountain forests: Review of indicators and value functions for model based multi-criteria decision analysis“, Ecol. Indic., Bd. 79, S. 391–409, Aug. 2017, doi: 10.1016/j.ecolind.2017.04.025.
- E. G. Brockerhoff et al., 2017, „Forest biodiversity, ecosystem functioning and the provision of ecosystem services“, Biodivers. Conserv., Bd. 26, Nr. 13, S. 3005–3035, Dez. 2017, doi: 10.1007/s10531-017-1453-2.
- H. Pretzsch, 2019, Grundlagen der Waldwachstumsforschung, 2. Aufl. Berlin: Springer, 2019. [Online]. Verfügbar unter: https://link.springer.com/book/10.1007/978-3-662-58155-1
- T. Riedel, P. Hennig, F. Kroiher, H. Polley, F. Schmitz, und F. Schwitzgebel, 2017, Die dritte Bundeswaldinventur BWI 2012, Inventur- und Auswertungsmethoden. Eberswalde: Thünen.
- C. E. Shannon, 1948, „The mathematical theory of communication“, in Shannon CE, Weaver W (eds): The mathematical theory of communication., Urbana: University of Illinois Press, 1948, S. 3–91.
- H. Wickham, M. Çetinkaya-Rundel, und G. Grolemund, 2023, R for Data Science: Import, Tidy, Transform, Visualize, and Model Data. O’Reilly Media, Inc.
- H. Wickham, M. Çetinkaya-Rundel, und G. Grolemund, 2024, R für Data Science: Daten importieren, bereinigen, umformen, modellieren und visualisieren. O’Reilly Media Inc.



